R을 통한 비구획분석: 실습
2019-03-04
Chapter 1 서론
약동학 분야에서 가장 간단하고도 객관적이며 널리 쓰이는 방법은 비구획분석 (Non-compartmental analysis, NCA)입니다. 미국의 FDA (Food and Drug Administration)를 비롯한 대부분의 규제기관에서는 NCA하는 소프트웨어를 규정하고 있지 않아, 상용 소프트웨어를 사용하지 않고 약동학적 지표를 구하는 것을 허용하고 있습니다. 따라서 무료로 누구나 사용할 수 있는 R 패키지를 사용하여 비구획분석을 통한 약동학적 주요 지표를 구할 수 있습니다.
1.1 설치
우선 R을 설치합니다. R은 아래 링크1에서 다운로드 받을 수 있습니다.
R을 실행한 후, 콘솔 창에서 비구획분석을 위한 패키지를 설치하는 방법은 다음과 같습니다. 홑따옴표 등의 인용 부호에 주의하세요.
install.packages('NonCompart')
install.packages('ncar')
install.packages('pkr')설치는 한번만 하면 되지만, 비구획분석을 위해서는 매 세션마다 패키지를 불러오기해야 합니다.
library(NonCompart)
library(ncar)
#library(pkr) # NonCompart::tblNCA() can be overwritten.아래 두 패키지는 비구획분석과는 관계없지만 자료 처리 혹은 그림 등을 그리는데 도움을 줍니다.
# install.packages(c('tidyverse', 'knitr'))
# 설치 안되어 있으면 맨앞의 #을 지우고 설치.
library(tidyverse) # For presentation only, dplyr, tidyr, ggplot2
library(knitr) # For reports도움이 필요할때는 맨 앞에 물음표를 붙여서 콘솔창에 입력하거나 help() 함수를 사용합니다.
?NonCompart
help(tblNCA)1.2 R에 대하여
R (R Core Team 2018)은 통계 소프트웨어 입니다. 굉장히 유용한 소프트웨어이지만 이에 대해 여기서 자세히 설명하긴 힘듭니다. R에 대한 많은 책들을 bookdown.org2에서 무료로 읽을 수 있습니다. Coursera3에서 무료 온라인 강의를 들을 수 있습니다.
1.3 자료 탐색
자료의 첫 10개 (Table 1.1) 혹은 마지막 10개 관찰값만 보고 싶으면 다음을 입력합니다. 대상자 번호가 첫 열에 나와있고 시간 순서대로 혈장에서 측정한 테오필린의 농도가 나와있습니다.
head(Theoph, n=10)
tail(Theoph, n=10)| Subject | Wt | Dose | Time | conc |
|---|---|---|---|---|
| 1 | 79.6 | 4.02 | 0.00 | 0.74 |
| 1 | 79.6 | 4.02 | 0.25 | 2.84 |
| 1 | 79.6 | 4.02 | 0.57 | 6.57 |
| 1 | 79.6 | 4.02 | 1.12 | 10.50 |
| 1 | 79.6 | 4.02 | 2.02 | 9.66 |
| 1 | 79.6 | 4.02 | 3.82 | 8.58 |
| 1 | 79.6 | 4.02 | 5.10 | 8.36 |
| 1 | 79.6 | 4.02 | 7.03 | 7.47 |
| 1 | 79.6 | 4.02 | 9.05 | 6.89 |
| 1 | 79.6 | 4.02 | 12.12 | 5.94 |
그림을 그려서 대략적인 자료의 모습을 파악합니다. (Figure 1.1)
ggplot(Theoph, aes(Time, conc, group = Subject, color = Subject)) +
geom_point(size = 4) +
geom_line(size = 1) +
theme_bw() +
labs(title = 'Oral Administration of Theoph (320 mg)',
x = 'Time (hour)', y = 'Concentration (ng/mL)')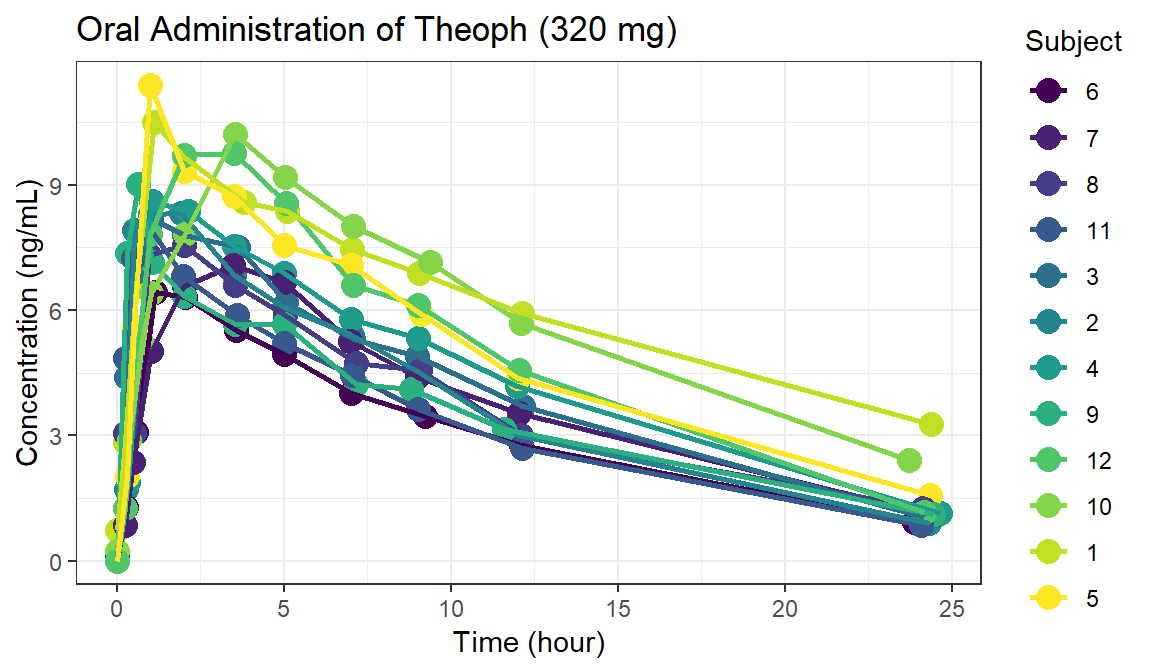
Figure 1.1: Concentration-time curves of oral administration of Theoph (N = 12)
1.4 자료 불러오기
read.csv() 함수를 사용해서 자료를 불러 옵니다.
엑셀 파일을 사용하는 경우 readxl 패키지를 설치한 후에 read_excel() 함수를 사용해서 불러올 수 있습니다.
다만 이 경우 tibble 형태로 자료가 변형되므로 as.data.frame()을 사용해서 데이타프레임으로 변형해주어야 합니다.
1.5 파라메터의 의미
비구획분석 시 여러 파라메터가 나오며 약어로 표현하는 경우가 많습니다. 또한 소프트웨어마다 약어가 상이하기 때문에 자주 그 의미를 찾아볼 필요가 있습니다. 콘솔창에 다음을 입력합니다.
?ncar::txtNCA()
ncar::RptCfgncar::RptCfg의 일부를 첨부합니다. (Table 1.2) PPTESTCD는 NonCompart 패키지에서 출력하는 파라메터 이름이며, CDISC SDTM PPTESTCD (Parameter Short Name)4와 같은 값입니다. WNL 열은 Certara Phoenix WinNonLin에서 구한 파라메터 이름입니다.
| PPTESTCD | Description (WNL) |
|---|---|
| b0 | Intercept (b0) |
| TLAG | Time Until First Nonzero Conc (Tlag) |
| MRTEVLST | MRT Extravasc to Last Nonzero Conc (MRTlast) |
| MRTEVIFO | MRT Extravasc Infinity Obs (MRTINF_obs) |
| MRTEVIFP | MRT Extravasc Infinity Pred (MRTINF_pred) |
| VZFO | Vz Obs by F (Vz_F_obs) |
| VZFP | Vz Pred by F (Vz_F_pred) |
| CLFO | Total CL Obs by F (Cl_F_obs) |
| CLFP | Total CL Pred by F (Cl_F_pred) |
| C0 | Initial Conc (C0) |
| AUCPBEO | AUC %Back Extrapolation Obs (AUC_.Back_Ext_obs) |
| AUCPBEP | AUC %Back Extrapolation Pred (AUC_.Back_Ext_pred) |
| CMAX | Max Conc (Cmax) |
| CMAXD | Max Conc Norm by Dose (Cmax_D) |
| TMAX | Time of CMAX (Tmax) |
| CLST | Last Nonzero Conc (Clast) |
| TLST | Time of Last Nonzero Conc (Tlast) |
| CLSTP | Last Nonzero Conc Pred (Clast_pred) |
| LAMZHL | Half-Life Lambda z (HL_Lambda_z) |
| LAMZ | Lambda z (Lambda_z) |
| LAMZLL | Lambda z Lower Limit (Lambda_z_lower) |
| LAMZUL | Lambda z Upper Limit (Lambda_z_upper) |
| LAMZNPT | Number of Points for Lambda z (No_points_lambda_z) |
| CORRXY | Correlation Between TimeX and Log ConcY (Corr_XY) |
| R2 | R Squared (Rsq) |
| R2ADJ | R Squared Adjusted (Rsq_adjusted) |
| AUCLST | AUC to Last Nonzero Conc (AUClast) |
| AUCALL | AUC All (AUCall) |
| AUCIFO | AUC Infinity Obs (AUCINF_obs) |
| AUCIFOD | AUC Infinity Obs Norm by Dose (AUCINF_D_obs) |
| AUCPEO | AUC %Extrapolation Obs (AUC_.Extrap_obs) |
| AUCIFP | AUC Infinity Pred (AUCINF_pred) |
| AUCIFPD | AUC Infinity Pred Norm by Dose (AUCINF_D_pred) |
| AUCPEP | AUC %Extrapolation Pred (AUC_.Extrap_pred) |
| AUMCLST | AUMC to Last Nonzero Conc (AUMClast) |
| AUMCIFO | AUMC Infinity Obs (AUMCINF_obs) |
| AUMCPEO | AUMC %Extrapolation Obs (AUMC_.Extrap_obs) |
| AUMCIFP | AUMC Infinity Pred (AUMCINF_pred) |
| AUMCPEP | AUMC % Extrapolation Pred (AUMC_.Extrap_pred) |
| MRTIVLST | MRT Intravasc to Last Nonzero Conc (MRTlast) |
| MRTIVIFO | MRT Intravasc Infinity Obs (MRTINF_obs) |
| MRTIVIFP | MRT Intravasc Infinity Pred (MRTINF_pred) |
| VZO | Vz Obs (Vz_obs) |
| VZP | Vz Pred (Vz_pred) |
| CLO | Total CL Obs (Cl_obs) |
| CLP | Total CL Pred (Cl_pred) |
| VSSO | Vol Dist Steady State Obs (Vss_obs) |
| VSSP | Vol Dist Steady State Pred (Vss_pred) |
References
Bae, Kyun-Seop. 2018b. NonCompart: Noncompartmental Analysis for Pharmacokinetic Data. https://CRAN.R-project.org/package=NonCompart.
Bae, Kyun-Seop. 2018a. Ncar: Noncompartmental Analysis for Pharmacokinetic Report. https://CRAN.R-project.org/package=ncar.
Bae, Kyun-Seop, and Jee Eun Lee. 2018. Pkr: Pharmacokinetics in R. https://CRAN.R-project.org/package=pkr.
R Core Team. 2018. R: A Language and Environment for Statistical Computing. Vienna, Austria: R Foundation for Statistical Computing. https://www.R-project.org/.
다음과 같이 CDISC note에 표시되어 있습니다. ‘Short name of the pharmacokinetic parameter. It can be used as a column name when converting a dataset from a vertical to a horizontal format. The value in PPTESTCD cannot be longer than 8 characters, nor can it start with a number (e.g., “1TEST”). PPTESTCD cannot contain characters other than letters, numbers, or underscores. Examples: “AUCALL”, “TMAX”, “CMAX”.’ https://wiki.cdisc.org/pages/viewpage.action?pageId=42309513↩